HDFS¶
Apuntes sin actualizar
Estos apuntes pertenecen al curso 21/22 y, por lo tanto, ya no se actualizan.
Puedes acceder a la última versión de esta sesión en https://aitor-medrano.github.io/iabd2223/hadoop/04hdfs.html.
Funcionamiento de HDFS¶
En la sesión anterior hemos estudiado los diferentes componentes que forman parte de HDFS: namenode y datanodes. En esta sesión veremos los procesos de lectura y escritura, aprenderemos a interactuar con HDFS mediante comandos, el uso de instantáneas y practicaremos con los formatos de datos más empleados en Hadoop, como son Avro y Parquet.
Procesos de lectura¶
Vamos a entender como fluyen los datos en un proceso de lectura entre el cliente y HDFS a partir de la siguiente imagen:
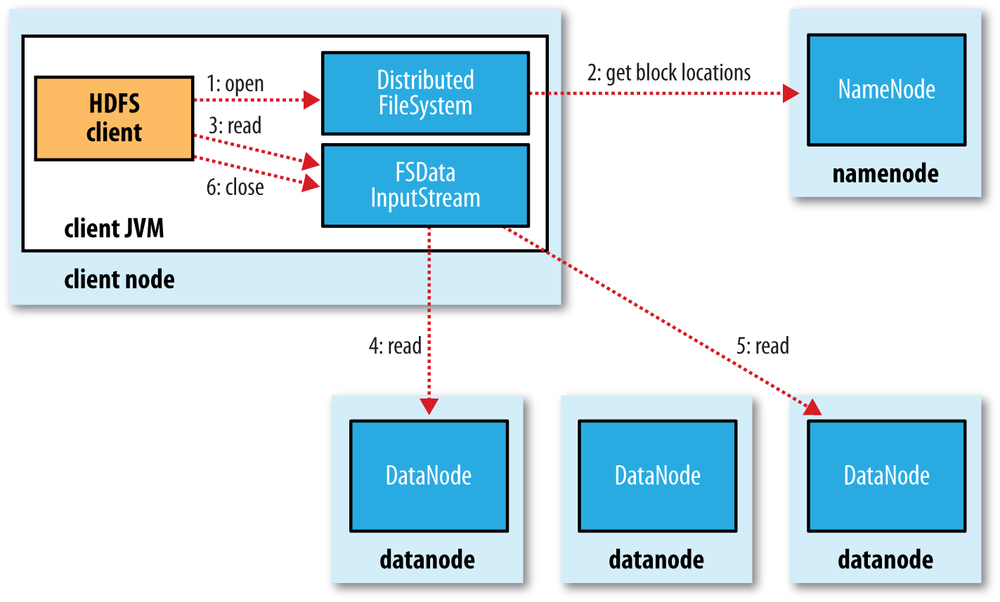
- El cliente abre el fichero que quiere leer mediante el método
open()del sistema de archivos distribuido. - Éste llama al namenode mediante una RPC (llamada a procedimiento remoto) el cual le indica la localización del primer bloque del fichero. Para cada bloque, el namenode devuelve la dirección de los datanodes que tienen una copia de ese bloque. Además, los datanodes se ordenan respecto a su proximidad con el cliente (depende de la topología de la red y despliegue en datacenter/rack/nodo). Si el cliente en sí es un datanode, la lectura la realizará desde su propio sistema local.
- El sistema de ficheros distribuido devuelve al cliente un FSDataInputStream (un flujo de entrada que soporta la búsqueda de ficheros), sobre el cual se invoca la lectura mediante el método
read(). Este flujo, que contiene las direcciones de los datanodes para los primeros bloques del fichero, conecta con el datanode más cercano para la lectura del primer bloque. - Los datos se leen desde el datanode con llamadas al método
read(). Cuando se haya leído el bloque completo, el flujo de entrada cerrará la conexión con el datanode actual y buscará el mejor datanode para el siguiente bloque. - Se repite el paso anterior (siempre de manera transparente para el cliente, el cual solo está leyendo datos desde un flujo de datos continuo).
- Cuando el cliente finaliza la lectura, cierra la conexión con el flujo de datos.
Durante la lectura, si el flujo encuentra un error al comunicarse con un datanode (o un error de checksum), intentará el proceso con el siguiente nodo más cercano (además, recordará los nodos que han fallado para no realizar reintentos en futuros bloques y/o informará de los bloque corruptos al namenode)
Namenode sin datos
Recordad que los datos nunca pasan por el namenode. El cliente que realiza la conexión con HDFS es el que hace las operaciones de lectura/escritura directamente con los datanodes. Este diseño permite que HDFS escale de manera adecuada, ya que el tráfico de los clientes se esparce por todos los datanodes de nuestro clúster.
Proceso de escritura¶
El proceso de escritura en HDFS sigue un planteamiento similar. Vamos a analizar la creación, escritura y cierre de un archivo con la siguiente imagen:
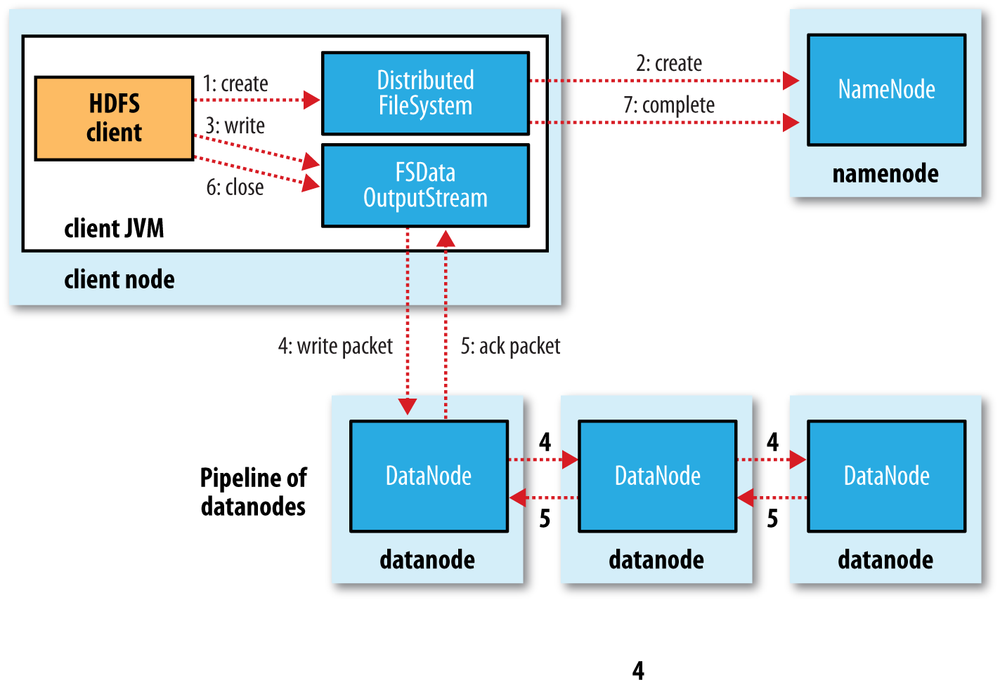
- El cliente crea el fichero mediante la llamada al método
create()del DistributedFileSystem. - Este realiza una llamada RPC al namenode para crear el fichero en el sistema de ficheros del namenode, sin ningún bloque asociado a él. El namenode realiza varias comprobaciones para asegurar que el fichero no existe previamente y que el usuario tiene los permisos necesarios para su creación. Tras ello, el namenode determina la forma en que va a dividir los datos en bloques y qué datanodes utilizará para almacenar los bloques.
- El DistributedFileSystem devuelve un FSDataOutputStream el cual gestiona la comunicación con los datanodes y el namenode para que el cliente comience a escribir los datos de cada bloque en el namenode apropiado.
- Conforme el cliente escribe los datos, el flujo obtiene del namenode una lista de datanodes candidatos para almacenar las réplicas. La lista de nodos forman un pipeline, de manera que si el factor de replicación es 3, habrá 3 nodos en el pipeline. El flujo envía los paquete al primer datanode del pipeline, el cual almacena cada paquete y los reenvía al segundo datanode del pipeline. Y así sucesivamente con el resto de nodos del pipeline.
- Cuando todos los nodos han confirmado la recepción y almacenamiento de los paquetes, envía un paquete de confirmación al flujo.
- Cuando el cliente finaliza con la escritura de los datos, cierra el flujo mediante el método
close()el cual libera los paquetes restantes al pipeline de datanodes y queda a la espera de recibir las confirmaciones. Una vez confirmado, le indica al namenode que la escritura se ha completado, informando de los bloques finales que conforman el fichero (puede que hayan cambiado respecto al paso 2 si ha habido algún error de escritura).
HDFS por dentro¶
HDFS utiliza de un conjunto de ficheros que gestionan los cambios que se producen en el clúster.
Primero entramos en $HADOOP_HOME/etc/hadoop y averiguamos la carpeta de datos que tenemos configurada en hdfs-site.xml para el namenode:
<property>
<name>dfs.name.dir</name>
<value>file:///opt/hadoop-data/hdfs/namenode</value>
</property>
Desde nuestro sistema de archivos, accedemos a dicha carpeta y vemos que existe una carpeta current que contendrá un conjunto de ficheros cuyos prefijos son:
edits_000NNN: histórico de cambios que se van produciendo.edits_inprogress_NNN: cambios actuales en memoria que no se han persistido.fsimagen_000NNN: snapshot en el tiempo del sistema de ficheros.
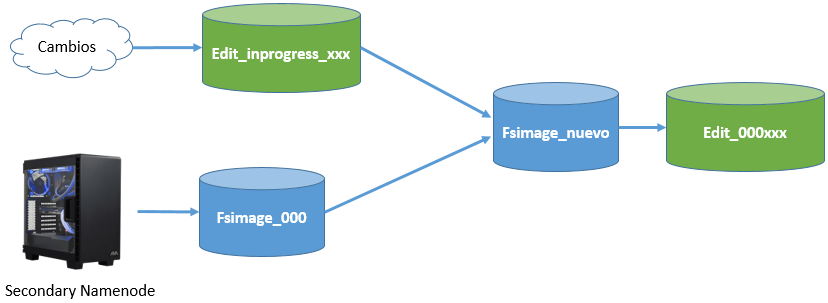
Al arrancar HDFS se carga en memoria el último fichero fsimage disponible junto con los edits que no han sido procesados. Mediante el secondary namenode, cuando se llena un bloque, se irán sincronizando los cambios que se producen en edits_inprogress creando un nuevo fsimage y un nuevo edits.
Así pues, cada vez que se reinicie el namenode, se realizará el merge de los archivos fsimage y edits log.
Trabajando con HDFS¶
Para interactuar con el almacenamiento desde un terminal, se utiliza el comando hdfs. Este comando admite un segundo parámetro con diferentes opciones.
Antes la duda, es recomendable consultar la documentación oficial
hdfs comando
hadoop fs
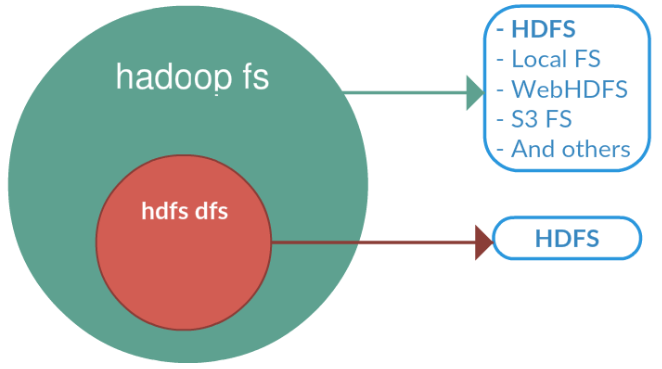
hadoop fs se relaciona con un sistema de archivos genérico que puede apuntar a cualquier sistema de archivos como local, HDFS, FTP, S3, etc. En versiones anteriores se utilizaba el comando hadoop dfs para acceder a HDFS, pero ya quedado obsoleto en favor de hdfs dfs.
En el caso concreto de interactuar con el sistema de ficheros de Hadoop se utiliza el comando dfs, el cual requiere de otro argumento (empezando con un guión) el cual será uno de los comandos Linux para interactuar con el shell. Podéis consultar la lista de comandos en la documentación oficial.
hdfs dfs -comandosLinux
Por ejemplo, para mostrar todos los archivos que tenemos en el raíz haríamos:
hdfs dfs -ls
Los comandos más utilizados son:
put: Coloca un archivo dentro de HDFSget: Recupera un archivo de HDFS y lo lleva a nuestro sistema host.cat/text/head/tail: Visualiza el contenido de un archivo.mkdir/rmdir: Crea / borra una carpeta.count: Cuenta el número de elementos (número de carpetas, ficheros, tamaño y ruta).cp/mv/rm: Copia / mueve-renombra / elimina un archivo.
Autoevaluación
¿Sabes qué realiza cada uno de los siguientes comandos?
hdfs dfs -mkdir /user/iabd/datos
hdfs dfs -put ejemplo.txt /user/iabd/datos/
hdfs dfs -put ejemplo.txt /user/iabd/datos/ejemploRenombrado.txt
hdfs dfs -ls datos
hdfs dfs -count datos
hdfs dfs -mv datos/ejemploRenombrado.txt /user/iabd/datos/otroNombre.json
hdfs dfs -get /datos/otroNombre.json /tmp
Bloques¶
A continuación vamos a ver cómo trabaja internamente HDFS con los bloques. Para el siguiente ejemplo, vamos a trabajar con un archivo que ocupe más de un bloque, como puede ser El registro de taxis amarillos de Nueva York - Enero 2020.
Comenzaremos creando un directorio dentro de HDFS llamado prueba-hdfs:
hdfs dfs -mkdir /user/iabd/prueba-hdfs
Una vez creado subimos el archivo con los taxis:
hdfs dfs -put yellow_tripdata_2020-01.csv /user/iabd/prueba-hdfs
Con el fichero subido nos vamos al interfaz gráfico de Hadoop (http://iabd-virtualbox:9870/explorer.html#/), localizamos el archivo y obtenemos el Block Pool ID del block information:
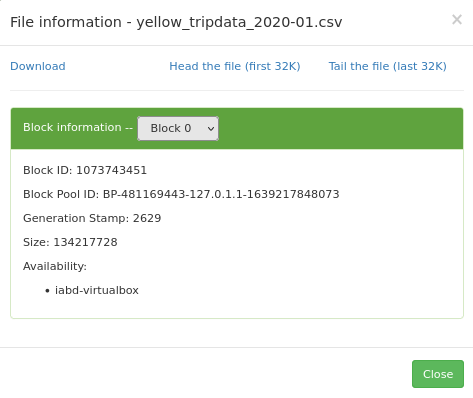
Si desplegamos el combo de block information, podremos ver cómo ha partido el archivo CSV en 5 bloques (566 MB que ocupa el fichero CSV / 128 del tamaño del bloque).
Así pues, con el código del Block Pool Id, podemos confirmar que debe existir el directorio current del datanode donde almacena la información nuestro servidor (en `/opt/hadoop-data/):
ls /opt/hadoop-data/hdfs/datanode/current/BP-481169443-127.0.1.1-1639217848073/current
Dentro de este subdirectorio existe otro finalized, donde Hadoop irá creando una estructura de subdirectorios subdir donde albergará los bloques de datos:
ls /opt/hadoop-data/hdfs/datanode/current/BP-481169443-127.0.1.1-1639217848073/current/finalized/subdir0
Una vez en este nivel, vamos a buscar el archivo que coincide con el block id poniéndole como prefijo blk_:
find -name blk_1073743451
En mi caso devuelve ./subdir6/blk_1073743451. De manera que ya podemos comprobar como el inicio del documento se encuentra en dicho archivo:
head /opt/hadoop-data/hdfs/datanode/current/BP-481169443-127.0.1.1-1639217848073/current/finalized/subdir0/subdir6/blk_1073743451
Administración¶
Algunas de las opciones más útiles para administrar HDFS son:
hdfs dfsadmin -report: Realiza un resumen del sistema HDFS, similar al que aparece en el interfaz web, donde podemos comprobar el estado de los diferentes nodos.hdfs fsck: Comprueba el estado del sistema de ficheros. Si queremos comprobar el estado de un determinado directorio, lo indicamos mediante un segundo parámetro:hdfs fsck /datos/pruebahdfs dfsadmin -printTopology: Muestra la topología, identificando los nodos que tenemos y al rack al que pertenece cada nodo.hdfs dfsadmin -listOpenFiles: Comprueba si hay algún fichero abierto.hdfs dfsadmin -safemode enter: Pone el sistema en modo seguro el cual evita la modificación de los recursos del sistema de archivos.
Snapshots¶
Mediante las snapshots podemos crear una instantánea que almacena cómo está en un determinado momento nuestro sistema de ficheros, a modo de copia de seguridad de los datos, para en un futuro poder realizar una recuperación.
El primer paso es activar el uso de snapshots, mediante el comando de administración indicando sobre qué carpeta vamos a habilitar su uso:
hdfs dfsadmin -allowSnapshot /user/iabd/datos
El siguiente paso es crear una snapshot, para ello se indica tanto la carpeta como un nombre para la captura (es un comando que se realiza sobre el sistema de archivos):
hdfs dfs -createSnapshot /user/iabd/datos snapshot1
Esta captura se creará dentro de una carpeta oculta dentro de la ruta indicada (en nuestro caso creará la carpeta /user/iabd/datos/.snapshot/snapshot1/ la cual contendrá la información de la instantánea).
A continuación, vamos a borrar uno de los archivo creados anteriormente y comprobar que ya no existe:
hdfs dfs -rm /user/iabd/datos/ejemplo.txt
hdfs dfs -ls /user/iabd/datos
Para comprobar el funcionamiento de los snapshots, vamos a recuperar el archivo desde la captura creada anteriormente.
hdfs dfs -cp \
/user/iabd/datos/.snapshot/snapshot1/ejemplo.txt \
/user/iabd/datos
Si queremos saber que carpetas soportan las instantáneas:
hdfs lsSnapshottableDir
Finalmente, si queremos deshabilitar las snapshots de una determinada carpeta, primero hemos de eliminarlas y luego deshabilitarlas:
hdfs dfs -deleteSnapshot /user/iabd/datos snapshot1
hdfs dfsadmin -disallowSnapshot /user/iabd/datos
HDFS UI¶
En la sesión anterior ya vimos que podíamos acceder al interfaz gráfico de Hadoop (http://iabd-virtualbox:9870/explorer.html#/) y navegar por las carpetas de HDFS.
Si intentamos crear una carpeta o eliminar algún archivo recibimos un mensaje del tipo Permission denied: user=dr.who, access=WRITE, inode="/":iabd:supergroup:drwxr-xr-x. Por defecto, los recursos via web los crea el usuario dr.who.
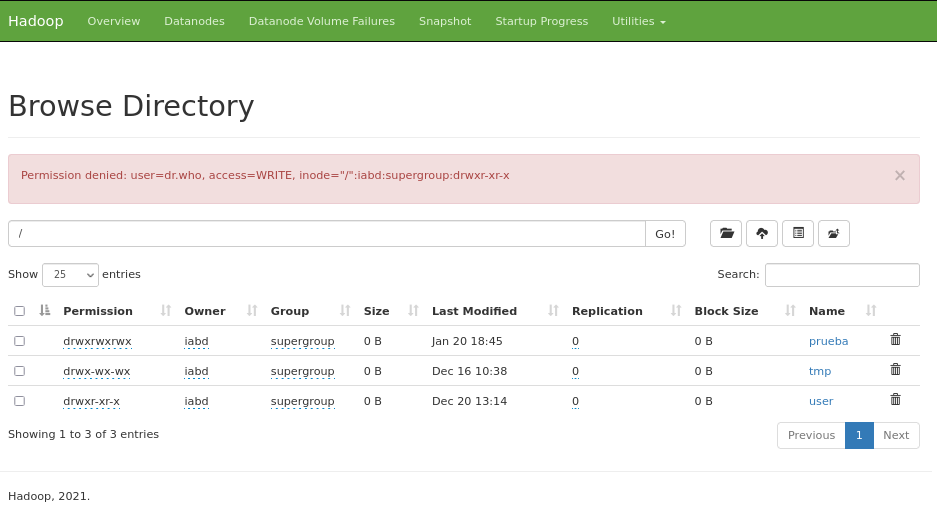
Si queremos habilitar los permisos para que desde este IU podamos crear/modificar/eliminar recursos, podemos cambiar permisos a la carpeta:
hdfs dfs -mkdir /user/iabd/pruebas
hdfs dfs -chmod 777 /user/iabd/pruebas
Si ahora accedemos al interfaz, sí que podremos trabajar con la carpeta pruebas via web, teniendo en cuenta que las operaciones las realiza el usuario dr.who que pertenece al grupo supergroup.
Otra posibilidad es modificar el archivo de configuración core-site.xml y añadir una propiedad para modificar el usuario estático:
<property>
<name>hadoop.http.staticuser.user</name>
<value>iabd</value>
</property>
Tras reiniciar Hadoop, ya podremos crear los recursos como el usuario iabd.
HDFS y Python¶
Para el acceso mediante Python a HDFS podemos utilizar la librería HdfsCLI (https://hdfscli.readthedocs.io/en/latest/).
Primero hemos de instalarla mediante pip:
pip install hdfs
Vamos a ver un sencillo ejemplo de lectura y escritura en HDFS:
from hdfs import InsecureClient
# Datos de conexión
HDFS_HOSTNAME = 'iabd-virtualbox'
HDFSCLI_PORT = 9870
HDFSCLI_CONNECTION_STRING = f'http://{HDFS_HOSTNAME}:{HDFSCLI_PORT}'
# En nuestro caso, al no usar Kerberos, creamos una conexión no segura
hdfs_client = InsecureClient(HDFSCLI_CONNECTION_STRING)
# Leemos el fichero de 'El quijote' que tenemos en HDFS
fichero = '/user/iabd/el_quijote.txt'
with hdfs_client.read(fichero) as reader:
texto = reader.read()
print(texto)
# Creamos una cadena con formato CSV y la almacenamos en HDFS
datos="nombre,apellidos\nAitor,Medrano\nPedro,Casas"
hdfs_client.write("/user/iabd/datos.csv", datos)
En el mundo real, los formatos de los archivos normalmente serán Avro y/o Parquet, y el acceso lo realizaremos en gran medida mediante la librería de Pandas.
Formatos de datos¶
En el primer bloque ya vimos una pequeña introducción a los diferentes formatos de datos.
Las propiedades que ha de tener un formato de datos son:
- independiente del lenguaje
- expresivo, con soporte para estructuras complejas y anidadas
- eficiente, rápido y reducido
- dinámico, de manera que los programas puedan procesar y definir nuevos tipos de datos.
- formato de fichero standalone y que permita dividirlo y comprimirlo.
Para que Hadoop pueda procesar documento, es imprescindible que el formato del fichero permita su división en fragmentos (splittable in chunks).
Si los clasificamos respecto al formato de almacenamiento tenemos:
- texto (más lentos, ocupan más pero son más expresivos y permiten su interoperabilidad): CSV, XML, JSON, etc...
- binarios (mejor rendimiento, ocupan menos, menos expresivos): Avro, Parquet, ORC, etc...
Si comparamos los formatos más empleados a partir de las propiedades descritas tenemos:
| Característica | CSV | XML / JSON | SequenceFile | Avro |
|---|---|---|---|---|
| Independencia del lenguaje | :thumbsup: | :thumbsup: | :fontawesome-regular-thumbs-down: | :thumbsup: |
| Expresivo | :fontawesome-regular-thumbs-down: | :thumbsup: | :thumbsup: | :thumbsup: |
| Eficiente | :fontawesome-regular-thumbs-down: | :fontawesome-regular-thumbs-down: | :thumbsup: | :thumbsup: |
| Dinámico | :thumbsup: | :thumbsup: | :fontawesome-regular-thumbs-down: | :thumbsup: |
| Standalone | :grey_question: | :thumbsup: | :fontawesome-regular-thumbs-down: | :thumbsup: |
| Dividible | :grey_question: | :grey_question: | :thumbsup: | :thumbsup: |
Las ventajas de elegir el formato correcto son:
- Mayor rendimiento en la lectura y/o escritura
- Ficheros trozeables (splittables)
- Soporte para esquemas que evolucionan
- Soporte para compresión de los datos (por ejemplo, mediante Snappy).
Filas vs Columnas¶
Los formatos con los que estamos más familiarizados, como son CSV o JSON, se basan en filas, donde cada registro se almacena en una fila o documento. Estos formatos son más lentos en ciertas consultas y su almacenamiento no es óptimo.
En un formato basado en columnas, cada fila almacena toda la información de una columna. Al basarse en columnas, ofrece mejor rendimiento para consultas de determinadas columnas y/o agregaciones, y el almacenamiento es más óptimo (como todos los datos de una columna son del mismo tipo, la compresión es mayor).
Supongamos que tenemos los siguientes datos:
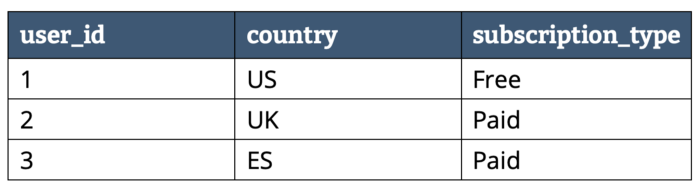
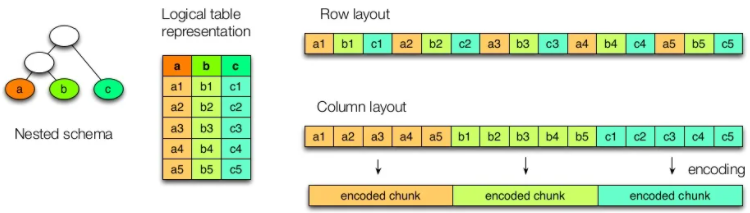
Dependiendo del almacenamiento en filas o columnas tendríamos la siguiente representación:

En un formato columnas los datos del mismo tipo se agrupan, lo que mejor el rendimiento de acceso y reduce el tamaño:
El artículo Apache Parquet: How to be a hero with the open-source columnar data format compara un formato basado en filas, como CSV, con uno basado en columnas como Parquet, en base al tiempo y el coste de su lectura en AWS (por ejemplo, AWS Athena cobra 5$ por cada TB escaneado):
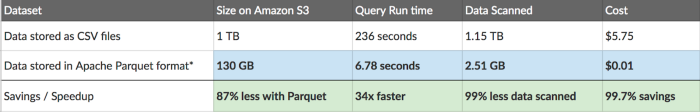
En la tabla podemos observar como 1TB de un fichero CSV en texto plano pasa a ocupar sólo 130GB mediante Parquet, lo que provoca que las posteriores consultas tarden menos y, en consecuencia, cuesten menos.
En la siguiente tabla comparamos un fichero CSV compuesto de cuatro columnas almacenado en S3 mediante tres formatos:

Queda claro que la elección del formato de los datos y la posibilidad de elegir el formato dependiendo de sus futuros casos de uso puede conllevar un importante ahorro en tiempo y costes.
Avro¶

Apache Avro es un formato de almacenamiento basado en filas para Hadoop, utilizado para la serialización de datos, ya que es más rápido y ocupa menos espacio que JSON, debido a que la serialización de los datos se realiza en un formato binario compacto.
Avro se basa en esquemas, los cuales se realizan mediante JSON para definir los tipos de datos y protocolos. Cuando los datos .avro son leídos siempre está presente el esquema con el que han sido escritos.
Schema¶
Los esquemas se componen de tipos primitivos (null, boolean, int, long, float, double, bytes, y string) y compuestos (record, enum, array, map, union, y fixed).
Un ejemplo de esquema podría ser:
{
"type" : "record",
"namespace" : "SeveroOchoa",
"name" : "Empleado",
"fields" : [
{ "name" : "Nombre" , "type" : "string" },
{ "name" : "Altura" , "type" : "float" }
{ "name" : "Edad" , "type" : "int" }
]
}
Python¶
Para poder serializar y deserializar documentos Avro mediante Python, previamente debemos instalar la librería avro:
pip install avro-python3
# o si utilizamos Anaconda
conda install -c conda-forge avro-python3
Vamos a realizar un ejemplo donde primero leemos un esquema de un archivo Avro, y con dicho esquema, escribiremos nuevos datos en un fichero. A continuación, abrimos el fichero escrito y leemos y mostramos los datos:
import avro
import copy
import json
from avro.datafile import DataFileReader, DataFileWriter
from avro.io import DatumReader, DatumWriter
# abrimos el fichero en modo binario y leemos el esquema
schema = avro.schema.parse(open("empleado.avsc", "rb").read())
# escribimos un fichero a partir del esquema leído
with open('empleados.avro', 'wb') as f:
writer = DataFileWriter(f, DatumWriter(), schema)
writer.append({"nombre": "Carlos", "altura": 180, "edad": 44})
writer.append({"nombre": "Juan", "altura": 175})
writer.close()
# abrimos el archivo creado, lo leemos y mostramos línea a línea
with open("empleados.avro", "rb") as f:
reader = DataFileReader(f, DatumReader())
# copiamos los metadatos del fichero leído
metadata = copy.deepcopy(reader.meta)
# obtenemos el schema del fichero leído
schemaFromFile = json.loads(metadata['avro.schema'])
# recuperamos los empleados
empleados = [empleado for empleado in reader]
reader.close()
print(f'Schema de empleado.avsc:\n {schema}')
print(f'Schema del fichero empleados.avro:\n {schemaFromFile}')
print(f'Empleados:\n {empleados}')
Schema de empleado.avsc:
{"type": "record", "name": "empleado", "namespace": "SeveroOchoa", "fields": [{"type": "string", "name": "nombre"}, {"type": "int", "name": "altura"}, {"type": ["null", "int"], "name": "edad", "default": null}]}
Schema del fichero empleados.avro:
{'type': 'record', 'name': 'empleado', 'namespace': 'SeveroOchoa', 'fields': [{'type': 'string', 'name': 'nombre'}, {'type': 'int', 'name': 'altura'}, {'type': ['null', 'int'], 'name': 'edad', 'default': None}]}
Empleados:
[{'nombre': 'Carlos', 'altura': 180, 'edad': 44}, {'nombre': 'Juan', 'altura': 175, 'edad': None}]
Fastavro¶
Para trabajar con Avro y grandes volúmenes de datos, se utiliza la librería Fastavro (https://github.com/fastavro/fastavro) la cual ofrece un rendimiento mucho mejor (en vez de estar codificada en Python puro, tiene algunos fragmentos realizados mediante Cython).
Primero, hemos de instalar la librería:
pip install fastavro
# o si utilizamos Anaconda
conda install -c conda-forge fastavro
Como podéis observar a continuación, hemos repetido el ejemplo y el código es muy similar:
import fastavro
import copy
import json
from fastavro import reader
# abrimos el fichero en modo binario y leemos el esquema
with open("empleado.avsc", "rb") as f:
schemaJSON = json.load(f)
schemaDict = fastavro.parse_schema(schemaJSON)
empleados = [{"nombre": "Carlos", "altura": 180, "edad": 44},
{"nombre": "Juan", "altura": 175}]
# escribimos un fichero a partir del esquema leído
with open('empleadosf.avro', 'wb') as f:
fastavro.writer(f, schemaDict, empleados)
# abrimos el archivo creado, lo leemos y mostramos línea a línea
with open("empleadosf.avro", "rb") as f:
reader = fastavro.reader(f)
# copiamos los metadatos del fichero leído
metadata = copy.deepcopy(reader.metadata)
# obtenemos el schema del fichero leído
schemaReader = copy.deepcopy(reader.writer_schema)
schemaFromFile = json.loads(metadata['avro.schema'])
# recuperamos los empleados
empleados = [empleado for empleado in reader]
print(f'Schema de empleado.avsc:\n {schemaDict}')
print(f'Schema del fichero empleadosf.avro:\n {schemaFromFile}')
print(f'Empleados:\n {empleados}')
Schema de empleado.avsc:
{'type': 'record', 'name': 'SeveroOchoa.empleado', 'fields': [{'name': 'nombre', 'type': 'string'}, {'name': 'altura', 'type': 'int'}, {'default': None, 'name': 'edad', 'type': ['null', 'int']}], '__fastavro_parsed': True, '__named_schemas': {'SeveroOchoa.empleado': {'type': 'record', 'name': 'SeveroOchoa.empleado', 'fields': [{'name': 'nombre', 'type': 'string'}, {'name': 'altura', 'type': 'int'}, {'default': None, 'name': 'edad', 'type': ['null', 'int']}]}}}
Schema del fichero empleadosf.avro:
{'type': 'record', 'name': 'SeveroOchoa.empleado', 'fields': [{'name': 'nombre', 'type': 'string'}, {'name': 'altura', 'type': 'int'}, {'default': None, 'name': 'edad', 'type': ['null', 'int']}]}
Empleados:
[{'nombre': 'Carlos', 'altura': 180, 'edad': 44}, {'nombre': 'Juan', 'altura': 175, 'edad': None}]
Fastavro y Pandas¶
Finalmente, vamos a realizar un último ejemplo con las dos librerías más utilizadas.
Vamos a leer un fichero CSV de ventas (que ya utilizamos en las sesiones de Pentaho) mediante Pandas, y tras limpiar los datos y quedarnos únicamente con las ventas de Alemania, almacenaremos el resultado del procesamiento en Avro.
import pandas as pd
from fastavro import writer, parse_schema
# Leemos el csv mediante pandas
df = pd.read_csv('pdi_sales.csv',sep=';')
# Limpiamos los datos (strip a los códigos postales) y nos quedamos con Alemania
df['Zip'] = df['Zip'].str.strip()
filtro = df.Country=="Germany"
df = df[filtro]
# 1. Definimos el esquema
schema = {
'name': 'Sales',
'namespace' : 'SeveroOchoa',
'type': 'record',
'fields': [
{'name': 'ProductID', 'type': 'int'},
{'name': 'Date', 'type': 'string'},
{'name': 'Zip', 'type': 'string'},
{'name': 'Units', 'type': 'int'},
{'name': 'Revenue', 'type': 'float'},
{'name': 'Country', 'type': 'string'}
]
}
schemaParseado = parse_schema(schema)
# 2. Convertimos el dataframe a una lista de diccionarios
records = df.to_dict('records')
# 3. Persistimos en un fichero avro
with open('sales.avro', 'wb') as f:
writer(f, schemaParseado, records)
import pandas as pd
from fastavro import parse_schema
from hdfs import InsecureClient
from hdfs.ext.avro import AvroWriter
from hdfs.ext.dataframe import write_dataframe
# 1. Nos conectamos a HDFS
HDFS_HOSTNAME = 'iabd-virtualbox'
HDFSCLI_PORT = 9870
HDFSCLI_CONNECTION_STRING = f'http://{HDFS_HOSTNAME}:{HDFSCLI_PORT}'
hdfs_client = InsecureClient(HDFSCLI_CONNECTION_STRING)
# 2. Leemos el dataframe
with hdfs_client.read('/user/iabd/pdi_sales.csv') as reader:
df = pd.read_csv(reader,sep=';')
# Limpiamos los datos (strip a los códigos postales) y nos quedamos con Alemania
df['Zip'] = df['Zip'].str.strip()
filtro = df.Country=="Germany"
df = df[filtro]
# 3. Definimos el esquema
schema = {
'name': 'Sales',
'namespace' : 'SeveroOchoa',
'type': 'record',
'fields': [
{'name': 'ProductID', 'type': 'int'},
{'name': 'Date', 'type': 'string'},
{'name': 'Zip', 'type': 'string'},
{'name': 'Units', 'type': 'int'},
{'name': 'Revenue', 'type': 'float'},
{'name': 'Country', 'type': 'string'}
]
}
schemaParseado = parse_schema(schema)
# 4a. Persistimos en un fichero avro dentro de HDFS mediante la extension AvroWriter de hdfs
with AvroWriter(hdfs_client, '/user/iabd/sales.avro', schemaParseado) as writer:
records = df.to_dict('records') # diccionario
for record in records:
writer.write(record)
# 4b. O directamente persistimos el dataframe mediante la extension write_dataframe de hdfs
write_dataframe(hdfs_client, '/user/iabd/sales2.avro', df) # infiere el esquema
write_dataframe(hdfs_client, '/user/iabd/sales3.avro', df, schema=schemaParseado)
Para el acceso HDFS hemos utilizados las extensiones Fastavro y Pandas de la librería HDFS del apartado anterior.
Comprimiendo los datos¶
¿Y sí comprimimos los datos para ocupen menos espacio en nuestro clúster y por tanto, nos cuesten menos dinero?
Fastavro soporta dos tipos de compresión: gzip (mediante el algoritmo deflate) y snappy. Snappy es una biblioteca de compresión y descompresión de datos de gran rendimiento que se utiliza con frecuencia en proyectos Big Data, la cual hemos de instalar previamente mediante pip install python-snappy.
Para indicar el tipo de compresión, únicamente hemos de añadir un parámetros extra con el algoritmo de compresión en la función/constructor de persistencia:
writer(f, schemaParseado, records, 'deflate')
with AvroWriter(hdfs_client, '/user/iabd/sales.avro', schemaParseado, 'snappy') as writer:
write_dataframe(hdfs_client, '/user/iabd/sales3.avro', df, schema=schemaParseado, codec='snappy')
Comparando algoritmos de compresión
Respecto a la compresión, sobre un fichero de 100GB, podemos considerar media si ronda los 50GB y alta si baja a los 40GB.
| Algoritmo | Velocidad | Compresión |
|---|---|---|
| Gzip | Media | Media |
| Bzip2 | Lenta | Alta |
| Snappy | Alta | Media |
Más que un tema de espacio, necesitamos que los procesos sean eficientes y por eso priman los algoritmos que son más rápidos. Si te interesa el tema, es muy interesante el artículo Data Compression in Hadoop.
Por ejemplo, si realizamos el ejemplo de Fast Avro y Pandas con acceso local obtenemos los siguientes tamaños:
- Sin compresión: 6,9 MiB
- Gzip: 1,9 MiB
- Snappy: 2,8 MiB
Parquet¶

Apache Parquet es un formato de almacenamiento basado en columnas para Hadoop, con soporte para todos los frameworks de procesamiento de datos, así como lenguajes de programación. De la misma forma que Avro, se trata de un formato de datos auto-descriptivo, de manera que embebe el esquema o estructura de los datos con los propios datos en sí. Parquet es idóneo para analizar datasets que contienen muchas columnas.
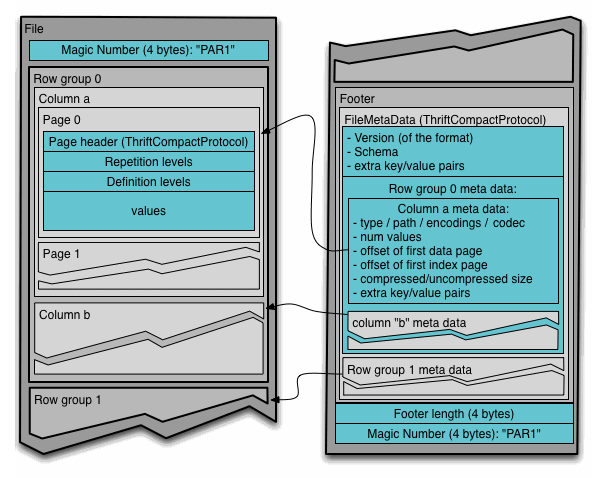
Cada fichero Parquet almacena los datos en binario organizados en grupos de filas. Para cada grupo de filas (row group), los valores de los datos se organizan en columnas, lo que facilita la compresión a nivel de columna.
La columna de metadatos de un fichero Parquet se almacena al final del fichero, lo que permite que las escrituras sean rápidas con una única pasada. Los metadatos pueden incluir información como los tipos de datos, esquemas de codificación/compresión, estadísticas, nombre de los elementos, etc...
Parquet y Python¶
Para interactuar con el formato Parquet mediante Python, la librería más utilizada es la que ofrece Apache Arrow, en concreto la librería PyArrow.
Así pues, la instalamos mediante pip:
pip install pyarrow
Apache Arrow usa un tipo de estructura denominada tabla para almacenar los datos bidimentsional (sería muy similar a un dataframe de Pandas). La documentación de PyArrow dispone de un libro de recetas con ejemplos con código para los diferentes casos de uso que se nos puedan plantear.
Vamos a simular el mismo ejemplo que hemos realizado previamente mediante Avro, y vamos a crear un fichero en formato JSON con empleados, y tras persistirlo en formato Parquet, lo vamos a recuperar:
import pyarrow.parquet as pq
import pyarrow as pa
# 1.- Definimos el esquema
schema = pa.schema([ ('nombre', pa.string()),
('altura', pa.int32()),
('edad', pa.int32()) ])
# 2.- Almacenamos los empleados por columnas
empleados = {"nombre": ["Carlos", "Juan"],
"altura": [180, 44],
"edad": [None, 34]}
# 3.- Creamos una tabla Arrow y la persistimos mediante Parquet
tabla = pa.Table.from_pydict(empleados, schema)
pq.write_table(tabla, 'empleados.parquet')
# 4.- Leemos el fichero generado
table2 = pq.read_table('empleados.parquet')
schemaFromFile = table2.schema
print(f'Schema del fichero empleados.parquet:\n{schemaFromFile}\n')
print(f'Tabla de Empleados:\n{table2}')
Para que pyarrow pueda leer los empleados como documentos JSON, a día de hoy sólo puede hacerlo leyendo documentos individuales almacenados en fichero:
Por lo tanto, creamos el fichero empleados.json con la siguiente información:
{ "nombre": "Carlos", "altura": 180, "edad": 44 }
{ "nombre": "Juan", "altura": 175 }
De manera que podemos leer los datos JSON y persistirlos en Parquet del siguiente modo:
import pyarrow.parquet as pq
import pyarrow as pa
from pyarrow import json
# 1.- Definimos el esquema
schema = pa.schema([ ('nombre', pa.string()),
('altura', pa.int32()),
('edad', pa.int32()) ])
# 2.- Leemos los empleados
tabla = json.read_json("empleados.json")
# 3.- Persistimos la tabla en Parquet
pq.write_table(tabla, 'empleados-json.parquet')
# 4.- Leemos el fichero generado
table2 = pq.read_table('empleados-json.parquet')
schemaFromFile = table2.schema
print(f'Schema del fichero empleados-json.parquet:\n{schemaFromFile}\n')
print(f'Tabla de Empleados:\n{table2}')
En ambos casos obtendríamos algo similar a:
Schema del fichero empleados.parquet:
nombre: string
altura: int32
edad: int32
Tabla de Empleados:
pyarrow.Table
nombre: string
altura: int32
edad: int32
----
nombre: [["Carlos","Juan"]]
altura: [[180,44]]
edad: [[null,34]]
Parquet y Pandas¶
En el caso del uso de Pandas el código todavía se simplifica más. Si reproducimos el mismo ejemplo que hemos realizado con Avro tenemos que los Dataframes ofrecen el método to_parquet para exportar a un fichero Parquet:
import pandas as pd
df = pd.read_csv('pdi_sales.csv',sep=';')
df['Zip'] = df['Zip'].str.strip()
filtro = df.Country=="Germany"
df = df[filtro]
# A partir de un DataFrame, persistimos los datos
df.to_parquet('sales.parquet')
Si quisiéramos almacenar el archivo directamente en HDFS, necesitamos indicarle a Pandas la dirección del sistema de archivos que tenemos configurado en core-site.xml:
<property>
<name>fs.defaultFS</name>
<value>hdfs://iabd-virtualbox:9000</value>
</property>
Así pues, únicamente necesitamos modificar el nombre del archivo donde serializamos los datos a Parquet:
df.to_parquet('hdfs://iabd-virtualbox:9000/sales.parquet')
Comparando tamaños¶
Si comparamos los tamaños de los archivos respecto al formato de datos empleado con únicamente las ventas de Alemania tendríamos:
ger_sales.csv: 9,7 MiBger_sales.avro: 6,9 MiBger_sales-gzip.avro: 1,9 MiBger_sales-snappy.avro: 2,8 MiB
ger_sales.parquet: 2,3 MiBger_sales-gzip.parquet: 1,6 MiBger_sales-snappy.parquet: 2,3 MiB
Hue¶
Hue (Hadoop User Experience) es una interfaz gráfica de código abierto basada en web para su uso con Apache Hadoop. Hue actúa como front-end para las aplicaciones que se ejecutan en el clúster, lo que permite interactuar con las aplicaciones mediante una interfaz más amigable que el interfaz de comandos.
En nuestra máquina virtual ya lo tenemos instalado y configurado para que funcione con HDFS y Hive.
La ruta de instalación es /opt/hue-4.10.0 y desde allí, arrancaremos Hue:
./build/env/bin/hue runserver
Tras arrancarlo, nos dirigimos a http://127.0.0.1:8000/y visualizaremos el formulario de entrada, el cual entraremos con el usuario iabd y la contraseña iabd:
Una vez dentro, por ejemplo, podemos visualizar e interactuar con HDFS:
Referencias¶
- Documentación de Apache Hadoop.
- Hadoop: The definitive Guide, 4th Ed - de Tom White - O'Reilly
- HDFS Commands, HDFS Permissions and HDFS Storage
- Introduction to Data Serialization in Apache Hadoop
- Handling Avro files in Python
- Native Hadoop file system (HDFS) connectivity in Python
Actividades¶
Para los siguientes ejercicios, copia el comando y/o haz una captura de pantalla donde se muestre el resultado de cada acción.
-
Explica paso a paso el proceso de lectura (indicando qué bloques y los datanodes empleados) que realiza HDFS si queremos leer el archivo
/logs/101213.log: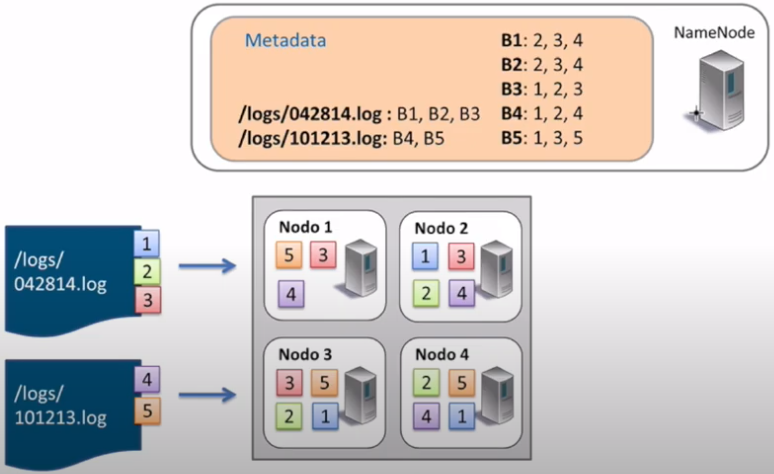
Proceso de lectura HDFS -
En este ejercicio vamos a practicar los comandos básicos de HDFS. Una vez arrancado Hadoop:
- Crea la carpeta
/user/iabd/ejercicios. - Sube el archivo
el_quijote.txta la carpeta creada. - Crea una copia en HDFS y llámala
el_quijote2.txt. - Recupera el principio del fichero
el_quijote2.txt. - Renombra
el_quijote2.txtael_quijote_copia.txt. - Adjunta una captura desde el interfaz web donde se vean ambos archivos.
- Vuelve al terminal y elimina la carpeta con los archivos contenidos mediante un único comando.
- Crea la carpeta
-
(opcional) Vamos a practicar los comandos de gestión de instantáneas y administración de HDFS. Para ello:
- Crea la carpeta
/user/iabd/instantaneas. - Habilita las snapshots sobre la carpeta creada.
- Sube el archivo
el_quijote.txta la carpeta creada. - Crea una copia en HDFS y llámala
el_quijote_snapshot.txt. - Crea una instantánea de la carpeta llamada
ss1. - Elimina ambos ficheros del quijote.
- Comprueba que la carpeta está vacía.
- Recupera desde
ssel archivoel_quijote.txt. - Crea una nueva instantánea de la carpeta llamada
ss2. - Muestra el contenido de la carpeta
/user/iabd/instantaneasasí como de sus snapshots.
- Crea la carpeta
-
(opcional) HDFS por dentro
- Accede al archivo de configuración
hdfs-site.xmly averigua la carpeta donde se almacena el namenode. - Muestra los archivos que contiene la carpeta
currentdentro del namenode - Comprueba el id del archivo
VERSION. - En los siguientes pasos vamos a realizar un checkpoint manual para sincronizar el sistema de ficheros. Para ello entramos en modo safe con el comando
hdfs dfsadmin -safemode enter, de manera que impedamos que se trabaje con el sistema de ficheros mientras lanzamos el checkpoint. - Comprueba mediante el interfaz gráfico que el modo seguro está activo (Safe mode is ON).
- Ahora realiza el checkpoint con el comando
hdfs dfsadmin -saveNamespace - Vuelve a entrar al modo normal (saliendo del modo seguro mediante
hdfs dfsadmin -safemode leave) - Accede a la carpeta del namenode y comprueba que los fsimage del namenode son iguales.
- Accede al archivo de configuración
-
Mediante Python, carga los datos de los taxis que hemos almacenado en HDFS en el apartado de Bloques y crea dentro de
/user/iabd/datoslos siguientes archivos con el formato adecuado:taxis.avro: la fecha (tpep_pickup_datetime), el VendorID y el coste de cada viaje (total_amount)- (opcional)
taxis.parquetcon los mismos atributos.